1.文章简介
| 文章题目 | CELF1 preferentially binds to exon-intron boundary and regulates alternative splicing in HeLa cells |
| 中文题目 | 在HeLa细胞中,CELF1倾向于结合在外显子-内含子的边界区域,并且影响可变剪接 |
| 期刊名 | BBA-Gene Regulatory Mechanisms, IF: 5.018 |
| 作者 | Heng Xia, Dong Chen, Qijia Wu, Gang Wu, Yanhong Zhou, Yi Zhang, Libin Zhang |
| 发表时间 | 2017年7月17日 |
| 实验材料 | HeLa |
| 测序平台 | Illumina HiSeq 2000, Illumina NextSeq 500 |
| 相关产品 | RIP-seq,RNA-seq |
2.研究背景
RNA结合蛋白(RBPs)参与到了转录后调控,包括RNA的可变剪接,RNA的空间折叠,从而导致RNA的表达量的变化。因此精确确定RBPs在RNA上的结合位置和结合特点是至关重要的。目前已经有成功解析RBP结合位点的方法,如CLIP-seq。但CLIP-seq也有很多的缺陷:如实验复杂,成功率低,文库的丰富度不够。和CLIP-seq比较类似的RIP-seq,虽然少了一些步骤,但获得的结果和CLIP-seq比较类似。最近有一项研究报道,使用DO-RIP-seq技术,也可以成功地获得RBPs的结合位点。而且能够获得更好的丰富度,这为RIP-seq分析RBPs的结合位点提供了方案和依据。
CELF1蛋白,也叫做CUGBP1.是一个经典的RNA结合蛋白。广泛地参与到RNA的可变剪接,mRNA的降解以及翻译。CELF1会倾向于结合U/G富集的序列。在本研究中,我们希望通过RIP-seq的方法,来获得CELF1在HeLa细胞中的结合特点,解析其对RNA可变剪接的影响。
3.研究内容
以HeLa细胞为细胞模型,使用RIP-seq技术,分析CELF1在RNA上的结合特点。同时分析了CELF1敲除后,对基因表达的影响,以及对可变剪接的影响。进而分析了这种影响是否是CELF1直接结合造成的。最后以LMO7为案例,说明了CELF1对可变剪接的影响,从而造成表达量的影响。
- CELF1的RIP-seq结果表明CELF1更倾向于结合在intron和3’UTR区域。
- CELF1的RIP-seq 会以可变剪接位点为中心形成结合峰,而且会富集GU-rich的基序。
- 对CELF1结合基因的功能分析,以及结合基因的验证结果。
- 在HeLa细胞中,CELF1会广泛调控pre-mRNA的可变剪接。
- CELF1通过直接结合,会调控LMO7基因的可变剪接。
4.研究方法
本研究主要使用了RIP-seq的技术方法,来获得CELF1在HeLa细胞中的结合特点,同时使用siCELF1的方法,获得CELF1直接和间接调控的基因,再结合CELF1的结合情况,从而更加清楚地获得了CELF1的调控模式。
5.研究成果
1. CELF1的RIP-seq结果表明CELF1更倾向于结合在intron和3’UTR区域。
使用RIP-seq技术,我们首先获得了CELF1在HeLa细胞中的结合特点。以IgG作为对照,我们从CELF1的IP样本中获得了2700万的测序片段,从IgG样本中获得了1200万的测序片段。使用tophat2方法对片段进行比对,然后分析其在基因组上的结合特点。发现CELF1更加倾向于结合在intron和3’UTR区域。
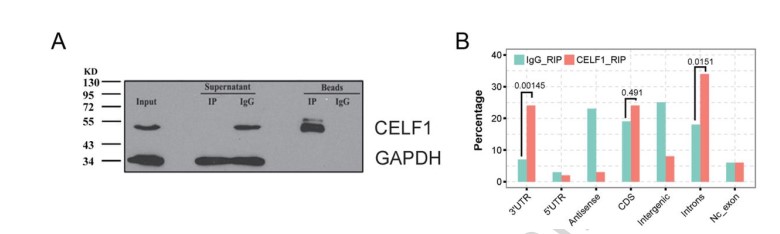
图1. RIP-seq实验展示了CELF1在HeLa细胞中的整体结合特点。(A)Western blot展示了CELF1 IP之后的结果图,说明IP实验是成功的。(B)展示了RIP-seq的reads在基因组上的分布特征,说明在3’UTR 和 intron区域是有富集的。
2.CELF1的RIP-seq 会以可变剪接位点为中心形成结合峰,而且会富集GU-rich的基序。
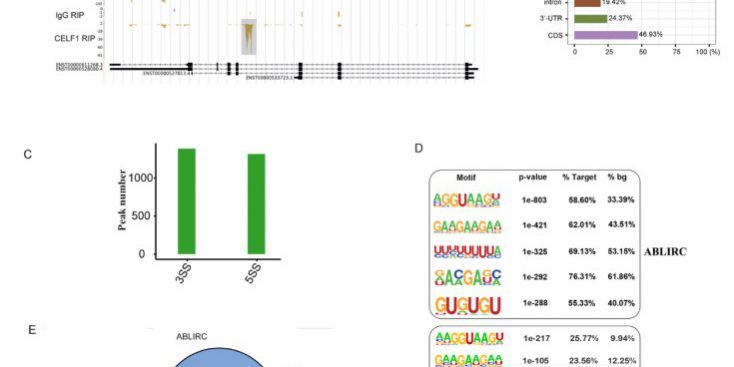
为了探究CELF1在HeLa细胞中的结合特点,本研究使用了ABLIRC的Peak Calling方法,对RIP-seq进行Peak分析。一共获得了15285个sense结合的peak,1385个antisense结合的peak。去掉IgG中出现的peak外,共获得了15542个结合的Peak,分布在5932个结合基因中。为了进一步分析RIP-seq数据的可靠性,本研究下载了已发表的CELF1的CLIP-seq数据,进行了一致性分析。在Peak和基因的层面,两者都有较好的一致性。因此RIP-seq的结果是可信的。CELF1的结合Peak大部分分布在CDS区域,而不是intron区域。因此我们推断CELF1可能会结合在exon-intron的边界区域,本研究把这样的peak归结为exon区域的Peak。分析CELF1在边界区域的结合情况,你有1319和1391个peak分布在3’SS和5’SS位置。本研究同时分析了CELF1的CLIP-seq Peak在此区域的结合情况,发现了类似的结果。
为了进一步分析CELF1的结合特点,本研究使用了Homer软件,对CELF1的结合Peak进行结合基序的富集性分析。发现除了经典的GU-rich基序之外,还富集到了AGGUAAG基序,以及U-rich的基序,分别对应5’SS和3’SS的剪接位点特点。为了进一步证明这种特点,本研究使用了另外一种Peak Calling方法:Piranha。也获得了相似的结果。

图2.CELF1 RIP-seq数据的Peak特点分析。(A)Peak在TMEM41B上的分布展示,一个显著的Peak落在了Intron区域。 (B)Peak分布区域的统计结果,在CDS,3’UTR和Intron都有较高的比例。(C)Peak的剪接位点的数量统计。(D)CELF1结合基序的结果展示,使用两种Peak Calling的方法,有相似的结果。(E)ABLIRC和Piranha两种方法获得Peak的重叠性分析。
3.对CELF1结合基因的功能分析,以及结合基因的验证结果
为了对CELF1 RIP-seq的结果进行验证,我们挑选了一些有CELF1 Peak的基因,进行RIP-PCR验证,通过图3A,可以发现,CELF1对这些基因确实有很强的结果。同时我们对CELF1结合的基因进行了功能聚类分析,通过图3B,发现CELF1结合基因富集在Spliceosome,按时CELF1可能通过调控Spliceosome相关的基因来调控基因的可变剪接。

图3. CELF1结合基因的验证,以及结合基因的功能分析。(A)使用RIP-PCR来验证CELF1结合的基因。(B)CELF1结合基因的功能聚类分析。
4.在HeLa细胞中,CELF1会广泛调控pre-mRNA的可变剪接。
通过CELF1的RIP-seq数据分析,研究发现CELF1可以通过结合pre-mRNA来影响可变剪接。因此研究继续进行了CELF1敲除后的RNA-seq实验,通过和NC比较,发现CELF1可以广泛调控HeLa细胞pre-mRNA的可变剪接。研究使用了两次RNA-seq的重复,分别获得了4631和19466个可变剪接事件。同时分析siCELF1和NC之间的差异,发现有1122个可变剪接事件发生了变化。而且有接近一半的可变剪接发生变化的基因,是CELF1结合的基因。同时,研究发现CELF1敲除之后,CELF1结合的exon的表达量发生了下调(图4D),也就是CELF1可以促进exon的跳跃(图4B)。这充分说明了CELF1对可变剪接的整体调控作用。

图4.CELF1基因对可变剪接的整体调控作用。(A)CELF1的Western Blot和RT-qPCR说明了CELF1的敲除效果是可信的。(B)受CELF1影响的可变剪接时间的柱状图展示。(C)CELF1结合基因和CELF1调控可变剪接基因的重叠性分析。(D)CELF1结合的exon的表达量变化展示。
5.CELF1通过直接结合,会调控LMO7基因的可变剪接。
本研究以LOM7为例,展示了CELF1对LMO7的可变剪接调控。研究发现,CELF1可以广泛地结合在LMO7上面,进一步分析,发现CELF1可以调控LMO7第16个外显子的可变剪接。通过进一步的实验表明,在CELF1敲除的情况下,LMO7的第16号外显子的跳跃确实发生了变化。因此我们可以断定,CELF1可以影响LMO7的可变剪接。

图5.CELF1对LMO7可变剪接的调控展示。(A)展示了CELF1在LMO7上的结果情况,同时发现对16号外显子有很明显的调控作用。(B)CELF1对LMO7第16号外显子调控的示意图,同时,使用RT-PCR和RT-qPCR进行了验证。(C)LMO7两种Isoform的测序结果展示。