1.文章简介
| 文章题目 | A Landscape of Pharmacogenomic Interactions in Cancer |
| 中文题目 | 癌症中的药物-基因组互作反应网络 |
| 期刊名 | Cell IF:28.71 |
| 发表时间 | 2016 |
| 实验材料 | 人类癌细胞系 |
| 相关产品 | 外显子组 |
2.研究背景
关于癌症过程中的基因组变异,科学家们已经开始积累大量的数据。通过对这些数据的分析和使用,可能让精准医疗更加系统化。
3.研究内容
2016年7月发表在Cell上的一篇文章中,研究者们比较了癌症患者肿瘤样本和癌细胞系中基因组变异的重叠程度,并检测了这些细胞系对抗癌药物的反应。通过分析获得的数据集,使得大规模预测抗癌药物的效果成为可能。
4研究方法
1、利用TCGA、ICGC和其他已发表的高通量测序数据分析人类肿瘤的变异特征。
2、利用1001种癌细胞系的高通量测序数据分析细胞系与肿瘤样本间的异同。
3、针对265种抗癌药物,检验990种癌细胞系的药物反应。
5研究结果
1,人类肿瘤的变异图谱
研究者们利用TCGA、ICGC和其他已发表的高通量测序数据(图1),绘制了人类肿瘤的变异图谱。图谱信息包括以下几类癌症功能事件(Cancer Functional Events, CFE):1、表现出定向选择特征的高突变率的癌症基因;2、反复发生的拷贝数差异变化;3、启动子区的超甲基化CpG位点。通过分析超过11000名病人的肿瘤样本信息,研究者们确定了1699种癌症特异CFE,并进一步合并为1273种泛癌症CFE。

图1、数据分析流程图
2. 肿瘤样本和癌细胞系中变异特征的保守性
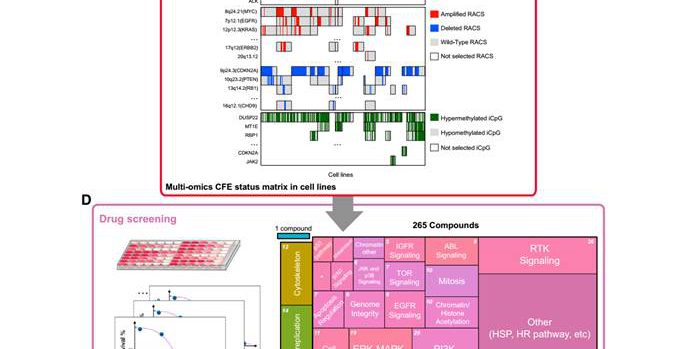
研究者们接下来对1001中癌细胞系的变异特征进行了分析,并与肿瘤样本的分析结果进行比较,发现两者之间有较好的一致性(图2)。对于肿瘤样本中鉴定的1273中泛癌症CFE,约84%在至少一种癌细胞系、约79%在至少3种癌细胞系中发生。其中,拷贝数变异在两者间一致性最高(100%),其次是超甲基化事件(89%)和癌症基因突变(64%)。同时,在检验了13条经典的癌相关信号通路后,研究者们发现同种类的肿瘤样本和癌细胞系之间同样具有高度的一致性(图3)。因此,尽管单一的癌细胞系的分子特征不能很好反映临床中的肿瘤特征,但通过多个癌细胞系的集合,研究者们可以较好的把握肿瘤中的变异、癌症通路变化和表观遗传变化等特征。

图2、癌症细胞系中的代表性癌症功能事件(CFE)

图3、癌细胞系和肿瘤样本的CFE特征比较
3. 癌症药物基因组学的互作模型
针对265种抗癌药物,研究者们检验了990种癌细胞系的药物反应。这些药物少数已进入临床使用,大部分仍处于早期开发或临床实验阶段。 针对获得的药物反应曲线进行分析,研究者们发现基因组变异特征往往能调控或预测药物敏感度,并由此鉴定了688种CFE-药物配对,其中540种配对为泛癌症的,174种为癌症特异的(图4)。

图4、抗癌药物敏感性的药物基因组互作模型
4、预测多种药物组合的抗癌效果
研究者们进一步对多种药物组合的抗癌效果进行预测(图6)。针对泛癌症的药物基因组互作,基因表达信息可较好预测药物反应;而对于癌症特异的药物,基因组变异特征可更好的用于预测药物反应。

图5、预测多种药物组合的抗癌效果
参考文献
Iorio,F., Knijnenburg,T.A., Vis,D.J., Bignell,G.R., Menden,M.P., Schubert,M., Aben,N., Gonçalves,E., Barthorpe,S., Lightfoot,H., et al. (2016) A Landscape of Pharmacogenomic Interactions in Cancer. Cell, 166, 740–754.