1.文章基本信息
| 文章题目 | Detection of 2019 novel coronavirus (2019-nCoV) by RT-PCR |
| 中文题目 | 应用RT-PCR 方法检测2019新型冠状病毒(2019-nCoV) |
| 期刊名 | Eurosurveillance |
| 作者 | Victor M Corman, Olfert Landt, Marco Kaiser, Richard Molenkamp, Adam Meijer, Daniel KW Chu, Tobias Bleicker, Sebastian Brünink, Julia Schneider, Marie Luisa Schmidt, Daphne GJC Mulders, Bart L Haagmans, Bas van der Veer, Sharon van den Brink, Lisa Wijsman, Gabriel Goderski, Jean-Louis Romette, Joanna Ellis, Maria Zambon, Malik Peiris, Herman Goossens, Chantal Reusken, Marion PG Koopmans, Christian Drosten |
| 发表时间 | 2020年1月23日 |
| 作者单位 | 1. 德国感染研究中心,2. 德国柏林Tib-Molbiol,3. 荷兰伊拉斯姆斯医学中心,4. 荷兰比尔托芬国家公共卫生与环境研究所,5. 香港大学,6. 法国马赛艾克斯马赛大学,7. 英格兰公共卫生部,8. 比利时安特卫普大学 |
摘要:
- 背景
最近新型冠状病毒(2019-nCoV)的持续爆发给公共卫生实验室带来了挑战。目前,这些实验室还无法分离病毒,且有证据表明,新型冠状病毒(2019-nCoV)疫情的传播范围远超出最初的想象,通过旅行者进行的国际传播已经出现。
- 目标
我们的目标是开发和研制可靠的诊断方法,用于公共卫生实验室检测新冠病毒。
- 方法
本文中,我们展示了一种新冠病毒检测诊断的工作流程。其引物和探针的设计依赖于新型冠状病毒与 SARS冠状病毒遗传上的相似序列和核酸合成技术。
- 结果
本方法能检测出新冠病毒(2019-nCoV),而且可以进一步区分新冠病毒与SARS病毒。通过学术实验室和公共卫生实验室之间的协调,我们完成了297例人类呼吸道病毒患者的临床标本的独家分析。对照材料来自于European Virus Archive Global (EVAg),一个欧洲联盟的基础设施项目。
- 结论
本次研究证明,通过协调国家和欧洲研究网络中的学术和公共实验室能产生巨大的反应能力。
引言:
世界卫生组织(WHO)中国国家办事处于2019年12月31日获悉湖北省武汉市出现不明原因肺炎病例[1]。2020年1月7日,中国官方正式宣布一种命名为2019-nCoV的新型冠状病毒为此次肺炎的病原体。1月10日,该病毒基因组序列随即通过公共在线资源公布(virological.org) (Wuhan-Hu-1, GenBank accession number MN908947[2]),另外4个基因组序列随后于1月12日在全球流感数据共享平台(GISAID)上公布。病毒基因组序列显示是一种和2003年爆发的 SARS病毒密切相关的病毒[3,4]。该物种由多种病毒组成,主要在亚洲和欧洲菊头蝠中检测到。
截至2020年1月20日,已向世卫组织通报了282例经实验室确诊的病例[5]。其中包括1月13日和17日在泰国以及1月15日在日本和1月19日在韩国确诊的病例,这些患者均为来自武汉的游客。在撰写本报告时,新型冠状病毒2019-nCoV的人际传播程度尚不清楚,但有证据表明存在一定程度的人与人之间的感染。
促进公共卫生干预的首要手段之一是可靠的实验室诊断。在急性呼吸道感染中,RT-PCR通常用于检测呼吸道分泌物中的病毒。通过协调公共实验室和学术实验室,我们已经论证了将基于实时RT-PCR的检测技术引入公共卫生实验室的可行性[6-12]。在所有这些检测中,病毒分离物可作为建立和控制检测, 及检测性能的首要底物。
在此次的新型冠状病毒事件中,国际公共卫生组织迄今为止尚未获得来自患者的病毒分离物或样本。本篇文章是在缺乏可用的病毒分离株或原始患者标本的情况下,建立并验证2019-nCoV筛查和特异性确认的诊断工作流程。该流程的设计和验证是基于新冠病毒(2019-nCoV)与2003年SARS-CoV较为紧密的遗传关系,并借助于核酸合成技术。
方法:
初步分析评价临床样本和冠状病毒细胞培养上清
由Charité和香港大学研究实验室提供了含有冠状病毒和其他呼吸道病毒的细胞培养上清。
2019年期间,我们从Charité医疗中心住院的病人身上采集了呼吸道样本,并由NxTAG呼吸道病理小组(Luminex, S´Hertogenbosch, The Netherlands)进行了检测。或者对以前公布通过 MERS-CoV upE检测方法确定的 MERS-CoV患者样本进行了检测[10]。其他更多样本分别来自比尔拖分 Rijksinstituut voor Volksgezondheid en Milieu (RIVM), 鹿特丹伊拉斯谟大学医学中心,英国伦敦公共卫生部和香港大学的生物库。所有收集的样本包括有病毒转运介质及没有病毒转运介质的痰液以及鼻咽拭子。
测试的粪便样本是含有提取自蝙蝠的SARS病毒相关的样本(已被证实GenBank accession Numbers),
- KC633203,Betacoronavirus BtCoV/Rhi_eur/BB98–98/BGR/2008;
- KC633204,Betacoronavirus BtCoV/Rhi_eur/BB98–92/BGR/2008;
- KC633201,Betacoronavirus BtCoV/Rhi_bla/BB98–22/BGR/2008;
- GU190221,Betacoronavirus Bat coronavirus BR98–19/BGR/2008;
- GU190222,Betacoronavirus Bat coronavirus BM98–01/BGR/2008;
- GU190223,Betacoronavirus Bat coronavirus BM98–13/BGR/2008.
这项研究中使用的所有合成RNA 都经过光学定量。
RNA提取
采用the MagNA Pure 96 system(Roche, Penzberg, Germany)从临床样本中提取RNA。
采用the viral RNA mini kit(QIAGEN, Hilden, Germany)从细胞培养上清液中提取病毒RNA。
实时逆转录PCR
一步RT-PCR反应体系(总体积25μL)
2X reaction buffer 12.5μL
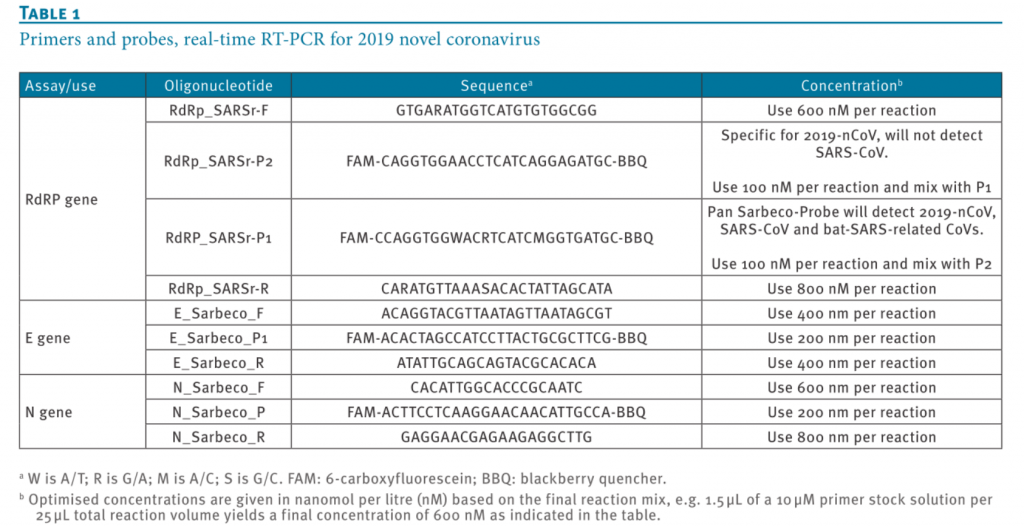
Forward primer 参照表1
Reverse primer 参照表1
Probe 参照表1
Superscript III 1μL
Dntp 0.4mmol/L(总浓度)
50 mM magnesium sulphate 0.4 μL (3.2mmol/L(总浓度))
RNA sample 5μL
总体积 25μL
所有的寡核苷酸都是由 Tib-molbiol公司(德国柏林)合成和提供的。
反应条件
逆转录:55°C反应 10分钟,95°C酶失活3分钟,PCR扩增45个循环(95°C变性15秒,58°C退火延伸30秒)。
参与的实验室使用罗氏公司的Light Cycler 480II或Applied Biosystems ViiA7仪器(Applied Biosystems,中国香港)。
表1:用于新冠病毒RT-PCR检测的引物和探针信息。
协议备选方案和应用说明
参与评估的实验室参考了相同寡核苷酸浓度和循环条件,只是反应试剂使用的是Thermo Fisher的一步转录反应液(TaqMan Fast Virus 1-Step)。
来自SARS病毒的RNA用于预期交叉反应检测,可以不用依赖于外部资源——2019新冠病毒RNA。
作为标准的工作流程,我们推荐E基因检测作为一线筛选工具,然后用RdRp 基因进行确认测试。 应用双色技术的RdRp基因检测,可以区分2019-nCoV 病毒 (两种探针均为阳性) ,SARS-CoV 病毒作为阳性对照。实验室可以选择只使用2019-nCoV 特异性探针进行 RdRp 基因检测。
伦理声明
用于诊断工作流程优化的样本是每个参与者对医学伦理规则达成一致后进行的。
结果:
在新冠病毒序列公开发布之前,我们依靠网络文献来开发类似SARS病毒的检测。因此我们假定认为SARS相关病毒在疾病爆发中出现。我们下载了截至2020年1月1日前GenBank中所有完整的和部分的(如果> 400 nt) SARS相关的病毒序列数据。729个数据经过手工检查,删除人工序列(实验室衍生的、合成的等)和重复序列后最终得到375个序列。这些序列经过比对分析后用于实验设计(图S1)。
图S1:SARS相关病毒序列比对结果——三种检测基因引物设计(分别为RdRp,E,N)
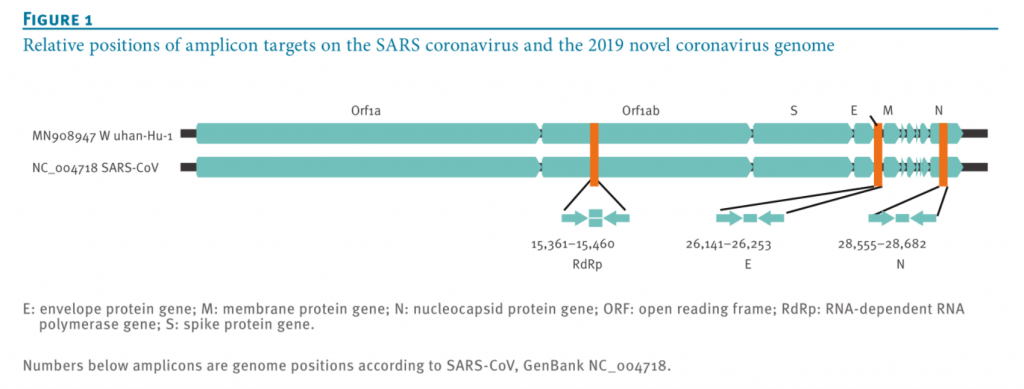
根据 virological.org 网站上发布的第一个新冠病毒序列,以及筛选序列和新冠病毒序列的吻合度,我们选择了三个检测位置(图1)。
图1:扩增靶标在SARS冠状病毒和新冠病毒基因组上的相对位置
通过对GISAID (https://www.gisaid.org)上独立发布的附加序列的互补比对,我们确认所设计引物与已知序列有较好的匹配率。引物结合区域与新冠病毒、SARS病毒以及选定的蝙蝠相关SARS病毒的序列比对结果(图2)。
图2:SARS相关冠状病毒(n=9)寡核苷酸结合区的部分比对展示
基于SARS冠状病毒的灵敏度检测
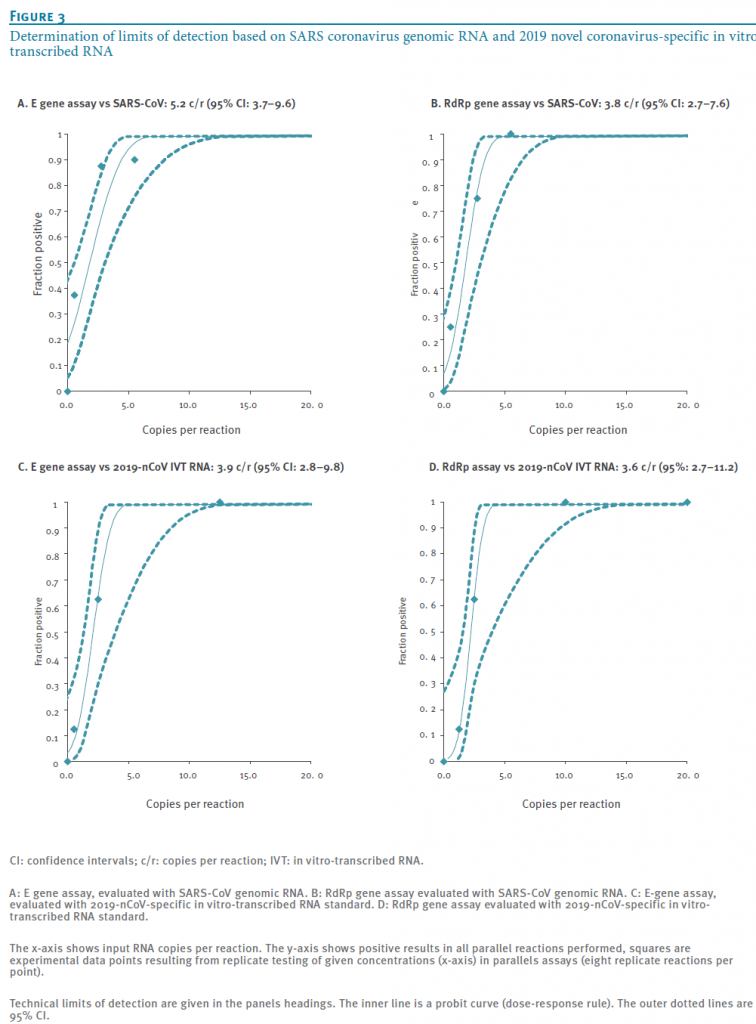
为了获得分析灵敏度的初步评估,我们使用含有SARS冠状病毒的纯化细胞培养上清液。上清液经过超滤,从大约20倍体积的细胞培养上清液中浓缩病毒。浓缩步骤同时降低背景核酸的相对浓度,如非病毒包装的病毒RNA。使用特定体外转录的RNA作为RT-PCR定量的标准,可参考Drosten等文章中描述[8]. 所有的试验都进行重复试验,以确定每个试验灵敏度终点的随机检测频率(图3,A和B)。
图3:基于SARS冠状病毒基因组RNA和新冠病毒特异性体外转录RNA检测限确定
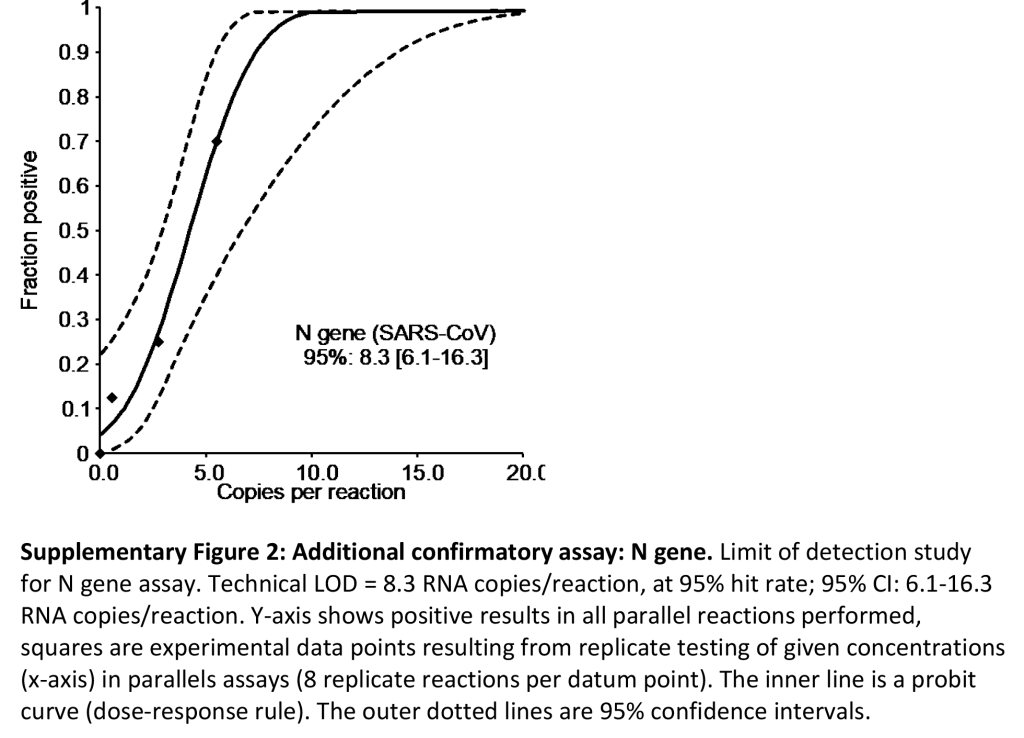
所有的检测方法都是高度敏感的,从E基因和RdRP基因检测获得最好的结果,所有的检测都是高度敏感的,这两个基因分别给出每个反应5.2和3.8个拷贝就可以获得95%检测可信度的结果。我们选择这两种化验方法以进一步评价。参与外部评价的实验室之一使用其他RT-PCR试剂(TaqMan Fast Virus 1-Step Master Mix)重复敏感性研究,结果是相同的(E基因:3.2 RNA拷贝/反应(95% CI: 2.2-6.8) ;RdRP: 3.7 RNA拷贝/反应(95% CI: 2.8-8.0))。值得注意的是,N基因测定也表现良好,但没有进行进一步的强化验证,因为它略微不那么敏感(图S2)。
图S2:N基因的附加验证实验—N基因的检测限研究
基于2019新型冠状病毒靶序列相同的体外转录RNA的敏感性检测
尽管两种方法都检测到2019-nCoV在寡核苷酸结合位点没有多态性(图2),我们还合成了与新冠病毒序列完全匹配的体外转录RNA,用于绝对定量和检测限(LOD)的研究。重复反应是在稀释试验中确定的检测终点附近的浓度下进行的。结果见图3C和3D,E基因(3.9个拷贝可获得95%检测可信度),RdRP基因(3.6拷贝可获得95%检测可信度)。根据泊松分布,当检测到一个RNA分子时,这些图接近2.9个拷贝可获得95%的命中率。
RdRp检测法区分2019-nCoV与SARS-CoV病毒
由于SARS冠状病毒RNA可以作为整个实验室过程的阳性对照,因此无需处理2019新冠病毒 RNA,我们设计了RdRp检测实验,包含两个探针:一条探针广泛的与SARS病毒和2019-新冠病毒反应,另一条探针只与2019-nCoV反应。通过限制稀释实验,我们证实,两种探针无论是单独使用还是联合使用,对每个病毒靶标都获得了相同的LOD值。特异性探针RdRP_SARSr-P2仅检测到新冠病毒 RNA转录本,而不能检测到SARS冠状病毒RNA。
来自蝙蝠的SARS相关病毒的检测
目前在早期报道的患者中,可能接触到共同的环境源,这意味着在病毒序列变异的情况下,存在着独立的人畜共患感染的可能[5]。我们使用E基因检测法检测了Drexler等人[13]和Muth等人[14]提供的六个蝙蝠粪便样本。可以检测到其它来自蝙蝠的SARS相关病毒。
呈阳性的粪便样本来自欧洲菊头蝠。在SARS相关冠状病毒中检测到这些系统发育离群病毒表明,所有亚洲病毒都有可能被检测到。从理论上讲,这将确保广泛的敏感性,可以检测到来自同一种动物宿主的多种变异病毒。
特异性测试:
化学稳定性
为了排除寡核苷酸之间的非特异性反应引起的人工荧光信号,所有检测方法均平行进行了120次检测,除引物外无其他核酸检出。这些反应都没有检测到任何阳性信号。
与其他冠状病毒的交叉反应
在所有三种试验中,包含所有地方性人类冠状病毒(HCoV)的细胞培养上清分别检测了 229E、 NL63、 OC43和 HKU1以及MERS-CoV (表2),对于不可培养的HCoV-HKU1,使用人气道培养的上清液。所有样本中的病毒RNA浓度由特定的实时RT-PCR和用于病毒载量绝对定量的体外转录RNA标准测定。其他未稀释(但未定量)的细胞培养上清检测(表2)。另外,这些样本被混合到阴性的人痰标本中。所有被测试的病毒或病毒制剂在任何检测中均未显示出反应性。
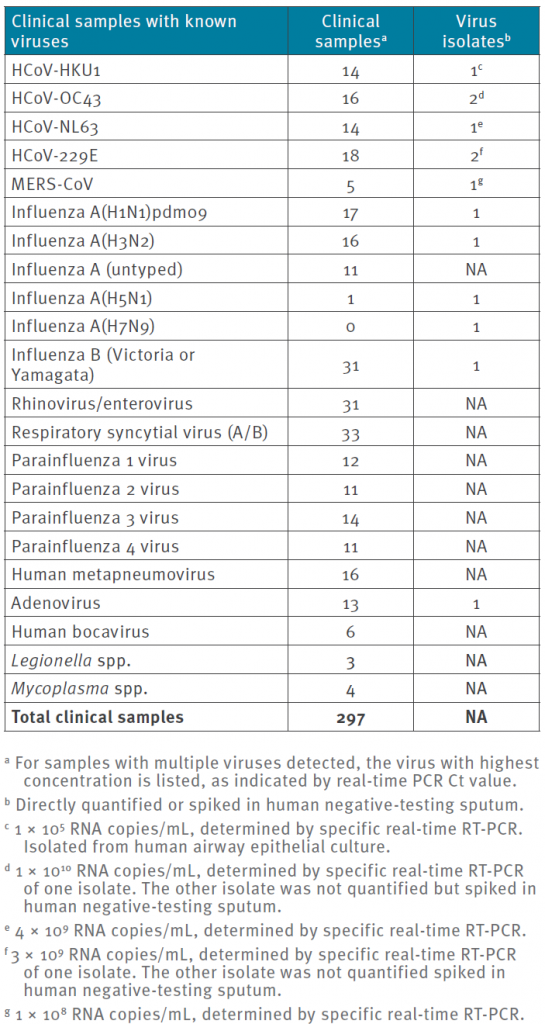
表2:新冠病毒E和RdRp基因检测中已知呼吸道病毒和细菌的临床样本和细胞培养制剂交叉反应试验(n=310)
2019年新型冠状病毒的排他性基于对其他呼吸道病毒的临床样本预检测阳性
利用E和RdRp基因检测方法,我们检测了来自五个提供诊断服务实验室(一个在德国,两个在荷兰,一个在香港,一个在英国)生物库的297个临床样本。我们从三个大学医疗中心(德国、荷兰、香港)选取了198个样本,这些医疗中心的病人来自普通病房、重症监护病房以及主要的儿科门诊。其余样本由国家公共卫生机构提供,主要由从业人员提供。这些样本含有极广泛的呼吸道病原体,反映了这些国家诊断实验室中病毒浓度的一般范围(表2)。总的来说,这项测试没有产生假阳性结果。在四个单独的测试反应中,初始反应微弱,但是用同样的测试方法重新测试时,结果都是阴性。这些信号与任何特定的病毒都没有关系,对于每一个最初出现阳性反应的病毒,都有其他样本含有更高浓度的相同病毒,但检测结果并非阳性。鉴于上述广泛的技术定量结果,我们得出结论,这种初始反应不是由于实时PCR试验的化学不稳定性引起的,而很可能是由于在本评估研究中引入了新的诊断试验和控制所引起的问题。
讨论:
在缺少自然来源的病毒基因组核酸的情况下,该团队建立了一种新型病毒诊断流程。与2003年的SARS冠状病毒相比,这种病毒的检测相对容易,这证明了对疾病生态学和病毒基因组多样性进行描述性研究具有巨大的价值[8,15-17]。
实时RT-PCR技术广泛应用于病毒学诊断。一旦发生公共卫生紧急事件,在新的诊断方法建立之前,专业的诊断实验室可以依赖这种成熟的技术提供常规检测服务。除了试剂、寡核苷酸和阳性对照的信息外,实验室还需要依靠检测方法的技术资格证明文件以及来自外部临床评价试验的数据。EVAg项目收集了学术研究中与病毒相关的试剂,有效地提供了参考RNA模板[18]。我们在2019新型冠状病毒爆发前从EVAg获得SARS冠状病毒 RNA;特定的产物——如文中所描述的检测所需的RNA转录本——也是首次从EVAg在2020年1月13日在线公布的目录中获得(https://www.european-virus-archive.com)。在2020年1月13日提供给世界卫生组织的第一版诊断方案中,包括了基于细胞培养材料和合成物结构的技术鉴定数据以及对75个临床样本进行排他性检测的结果。得益于实验室非正式网络的高效协作,这些数据在1周内得到了扩充,包括基于自然感染的临床样本中各种呼吸道病原体的检测结果。体外诊断试验监管资格的比较评估研究,对于生物组织、法律实施和后勤来说,可能需要几个月的时间,而且通常是在疫情高峰消退之后。近年来,为应对国际卫生危机而建立的国家和欧洲研究网络使目前的部署和评估工作的速度和成效得以提高,显示出通过学术和公共实验室的协调行动可以释放出巨大的应对能力[18-22]。实验室所具有的这种能力不仅能够对公共卫生干预措施提供直接的支持,而且能够确保在快速临床研究反应期间招募患者。
参考文献:
1. World Health Organization (WHO). Coronavirus. Geneva: WHO; 2020 [Accessed 21 Jan 2020]. Available from: https://www.who.int/health-topics/coronavirus
2. Zhang Y-Z. Novel 2019 coronavirus genome. Virological. [Accessed 21 Jan 2020]. Available from: http://virological.org/t/novel-2019-coronavirus-genome/319
3. de Groot RJ, Baker SC, Baric R, Enjuanes L, Gorbalenya AE, Holmes KV, et al. Family Coronaviridae. In: King AMQ, Adams MJ, Carstens EB, Lefkowitz EJ. Virus taxonomy: classification and nomenclature of viruses: ninth report of the International Committee on Taxonomy of Viruses. London; Waltham: Academic Press; 2012. p. 806-20.
4. Peiris JS, Yuen KY, Osterhaus AD, Stöhr K. The severe acute respiratory syndrome. N Engl J Med. 2003;349(25):2431-41. 10.1056/NEJMra032498
5. World Health Organization. (WHO. Novel Coronavirus (2019-nCoV). Situation report – 1. Geneva: WHO; 21 Jan 2020. Available from: https://www.who.int/docs/default-source/coronaviruse/situation-reports/20200121-sitrep-1-2019-ncov.pdf
6. Abbott A. SARS testing: First past the post. Nature. 2003;423(6936):114. 10.1038/423114a
7. Corman VM, Müller MA, Costabel U, Timm J, Binger T, Meyer B, et al. Assays for laboratory confirmation of novel human coronavirus (hCoV-EMC) infections. Euro Surveill. 2012;17(49):20334. 10.2807/ese.17.49.20334-en
8. Drosten C, Günther S, Preiser W, van der Werf S, Brodt HR, Becker S, et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med. 2003;348(20):1967-76. 10.1056/NEJMoa030747
9. Corman VM, Eickmann M, Landt O, Bleicker T, Brünink S, Eschbach-Bludau M, et al. Specific detection by real-time reverse-transcription PCR assays of a novel avian influenza A(H7N9) strain associated with human spillover infections in China. Euro Surveill. 2013;18(16):20461.
10. Corman VM, Eckerle I, Bleicker T, Zaki A, Landt O, Eschbach-Bludau M, et al. Detection of a novel human coronavirus by real-time reverse-transcription polymerase chain reaction. Euro Surveill. 2012;17(39):20285. 10.2807/ese.17.39.20285-en
11. Panning M, Charrel RN, Donoso Mantke O, Landt O, Niedrig M, Drosten C. Coordinated implementation of chikungunya virus reverse transcription-PCR. Emerg Infect Dis. 2009;15(3):469-71. 10.3201/eid1503.081104
12. Corman VM, Rasche A, Baronti C, Aldabbagh S, Cadar D, Reusken CB, et al. Assay optimization for molecular detection of Zika virus. Bull World Health Organ. 2016;94(12):880-92. 10.2471/BLT.16.175950
13. Drexler JF, Gloza-Rausch F, Glende J, Corman VM, Muth D, Goettsche M, et al. Genomic characterization of severe acute respiratory syndrome-related coronavirus in European bats and classification of coronaviruses based on partial RNA-dependent RNA polymerase gene sequences. J Virol. 2010;84(21):11336-49. 10.1128/JVI.00650-10
14. Muth D, Corman VM, Roth H, Binger T, Dijkman R, Gottula LT, et al. Attenuation of replication by a 29 nucleotide deletion in SARS-coronavirus acquired during the early stages of human-to-human transmission. Sci Rep. 2018;8(1):15177. 10.1038/s41598-018-33487-8
15. Corman VM, Muth D, Niemeyer D, Drosten C. Hosts and sources of endemic human coronaviruses.Adv Virus Res. 2018;100:163-88. 10.1016/bs.aivir.2018.01.001
16. Drexler JF, Corman VM, Drosten C. Ecology, evolution and classification of bat coronaviruses in the aftermath of SARS. Antiviral Res. 2014;101:45-56. 10.1016/j.antiviral.2013.10.013
17. Cui J, Li F, Shi ZL. Origin and evolution of pathogenic coronaviruses. Nat Rev Microbiol. 2019;17(3):181-92. 10.1038/s41579-018-0118-9
18. Romette JL, Prat CM, Gould EA, de Lamballerie X, Charrel R, Coutard B, et al. The European Virus Archive goes global: A growing resource for research. Antiviral Res. 2018;158:127-34. 10.1016/j.antiviral.2018.07.017
19. Alleweldt F, Kara S, Osinski A, Van Baal P, Kellerborg K, Aarestrup FM, et al. Developing a framework to assess the costeffectiveness of COMPARE – a global platform for the exchange of sequence-based pathogen data. Rev Sci Tech. 2017;36(1):311-22. 10.20506/rst.36.1.2631
20. Domingo C, Ellerbrok H, Koopmans M, Nitsche A, Leitmeyer K, Charrel RN, et al. Need for additional capacity and improved capability for molecular detection of yellow fever virus in European Expert Laboratories: External Quality Assessment, March 2018. Euro Surveill. 2018;23(28):1800341. 10.2807/1560-7917.ES.2018.23.28.1800341
21. Pas SD, Patel P, Reusken C, Domingo C, Corman VM, Drosten C, et al. First international external quality assessment of molecular diagnostics for Mers-CoV. J Clin Virol. 2015;69:81-5. 10.1016/j.jcv.2015.05.022
22. Gobat N, Amuasi J, Yazdanpanah Y, Sigfid L, Davies H, Byrne JP, et al. Advancing preparedness for clinical research during infectious disease epidemics. ERJ Open Res. 2019;5(2):00227-2018. 10.1183/23120541.00227-2018
因为译者能力和知识范围的局限,本文的翻译难免有不到位和错误的地方,欢迎各位读者留言讨论、批评和指正。译者会根据读者的讨论和建议及时更新。本系列翻译旨在尽一点微薄之力支持大众的“抗疫”行动和国家的“抗疫”决策。
译者 | 余凤云
编辑 | 日月明
免责申明| 仅供学习交流