1.文章简介
| 文章题目 | Interconnected microbiomes and resistomes in low-income human habitats |
| 中文题目 | 耐药基因组与微生物在低收入人群中的关联研究 |
| 期刊名 | Nature IF:41.456 |
| 发表时间 | 2016.05 |
| 单位 | Center for Genome Sciences and Systems Biology, Washington University School of Medicine |
| 实验材料 | Human faecal and environmental samples |
| 测序平台 | Illumina HiSeq and Nextseq |
| 相关产品 | Metagenomics |
2.研究背景
抗生素耐药感染每年造成成千上万人的死亡;
耐药基因在微生物和病原菌之前的传递加剧这一尖锐问题;
研究耐药基因组的分布特点以及其潜在的不同种群能够有助于辨别微生物和抗性基因;
大部分耐药基因组学研究集中在工业化社会或者远古时代背景,而地球上大部分人生活在低等或中等收入国家,其中八亿六千三百万人生活在贫民窟;对这一人群进行耐药基因的研究将意义重大。
3.研究方法
利用宏基因组学在低等收入人群中比较研究微生物种群和相关联的耐药基因的系统进化结构。
研究思路和亮点
通过分析两年中来自27个家族中的115个人类的263个样本,以及与209个环境样本进行比较。联合使用16S测序、高通量功能宏基因组筛选以及全宏基因组鸟枪法测序技术,比较微生物种群和相关耐药基因组的系统发生结构。
4.研究成果
1.耐药基因组与不同生态系统的系统发生的相关性
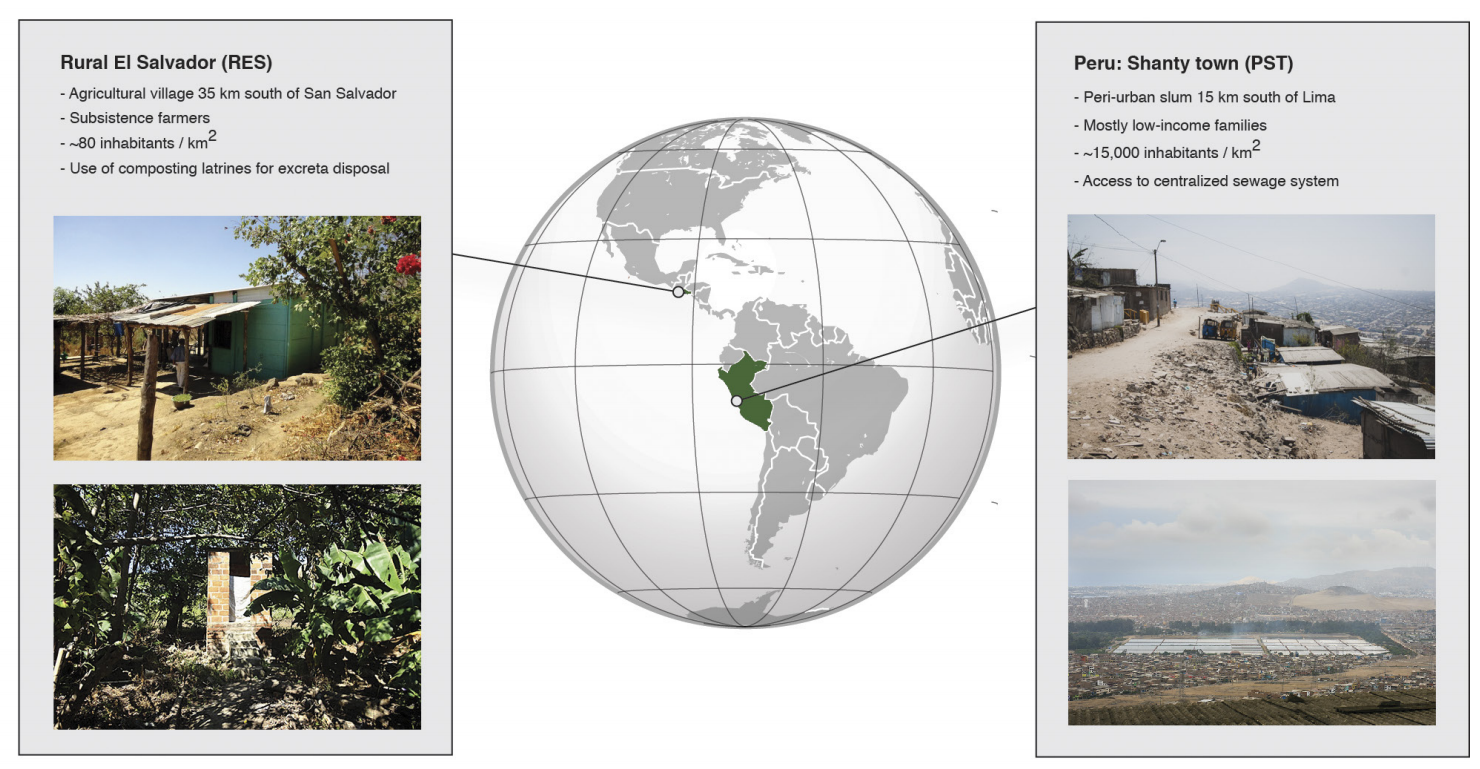
利用大肠杆菌进行小插入片段宏基因组表达文库构建,对来自农村生活的农民群体(RES)和近城市的棚户区居民群体(PST)的51个人类粪便和45个环境样本进行17个抗生素抗性研究。测序和基因注释鉴定出1100个特异的抗生素抗性编码基因,包含除美罗培南外的所有抗生素抗性基因,且氨基酸序列达到100%一致。其中,121个蛋白为新发现(小于60%氨基酸与NCBI nr离任何蛋白质序列一致),其中大多数(72%)为预测的抗生素修饰蛋白,包含57个Aβ-内酰胺酶类型。RES公厕文库产出了大多数的新发现的蛋白(46%)。
为了研究RES和PST耐药基因组的多样性和丰度,进一步对191个人类粪便和94个环境样本进行了全基因组的鸟枪法测序,产出344Gb数据。利用ShortBRED和定制的耐药基因数据库(囊括本研究所有基因)对所有测序的宏基因组中的耐药基因进行定量。以人类粪便为Input,RES和PST人类种群和环境耐药基因组在生态梯度上相关联,解释了22.4%的耐药基因组突变。Procrustes分析确认了抗生素耐药在群落组成中是显著关联的,而非在不同群体中随机分布((Bray–Curtis, M2 = 0.360, P < 0.001; Extended Data Fig. 2c)。
2.RES/PST的人类粪便微生物比较
通过比较RES和PST与已发表的微生物学数据,包含USA,Malawi,Venezuela和Peru。RES微生物与乡村农活工作者和hunter聚集人群聚类,而PST 介于这一人群与城市工业化群体之间(adonis R2= 23.8%, P < 0.001, weighted UniFrac; Fig. 1a and Extended Data Fig. 3)。为了评估抗生素耐药是否与工业化程度相关,对RES、PST与美国工业化群体、传统hunter-gather以及乡村耕种者群体相比较,结果发现PST群体中,个人携带抗生素抗性蛋白最高。
3.RES群体与环境微生物
乡村耕作和不太完善的排泄物管理方式会加速耐药基因的频繁接触,很有可能促进耐药基因的交换。通过principal coordinate analysis (PCoA),对系统发生进行了研究。发现人类粪便微生物在系统发生组成上比公厕、土壤和水中微生物同源性更高,这可能与环境样本的多样化和可变因素较多相关。并且,人类粪便与包含鸡排泄物的土壤拥有10个相同的耐药基因,说明鸡排泄物可能充当了人类排泄物与土壤微生物的一种桥梁作用。
4.PST人与污水
为了检测污水处理对耐药基因组的影响,对PST居民居住地附近的街道排污口以及本地的现代污水处理工厂(WWTP)的进水口和出水口进行取样。将它们与PST居民粪便样品比较研究其系统发生组成和耐药基因组。虽然街道排污口在距离上与居民区较近,但是其微生物组成与WWTP进水口更为接近。PST人类垃圾在通过污水处理过程中出现了显著的组成改变,大大降低了其余人类粪便微生物组成的相似度。
同样,污水耐药基因组也与微生物组成一样出现类似的改变,随着污水处理进程,街道排污口和WWTP进水口与人类粪便耐药基因组内容较为接近。虽然WWTP排水口有着最高的生态多样性(Fig. 3c),但是每个样本中的耐药基因数却是最少的(Fig. 3d)。相反,寄到排出口和WWTP进水口有着相同数量的生态多样性,其每个样本中的耐药基因数量与PST人类粪便相比也是更多。药物排泄抗生素抵抗机制在PST人粪便中显著高于排污口耐药基因组。相反,污水中富集了氨基葡糖苷乙酰转移酶,Dβ乙酰胺酶和抗性二氢叶酸还原酶。
与预期一致,人类粪便对于WWTP出水口的系统多态性组成贡献要显著低于街道排污设施或者WWTP进水口。有趣的是,这一差异在耐药基因组层面来讲却并不显著。虽然排污处理能够成功的降低总的耐药基因组以及人类粪便微生物,但是粪便原始耐药基因的生存在污水排除后可能重新导入到自然环境中。
5.大量AR和HGT在微生物群体间发生
大量的抗生素抗性基因在人类、动物和环境中均有检测到,其中HGT促进了细菌基因组和细菌宿主间的基因传递。通过对公认的移动遗传原件(MGEs)和多药抗性基因簇(MDRCs)注释,发现MGEs或MDRCs确实对于抗性基因在环境和病原菌间的传递启着一定的推动作用。
原文出处:
Pehrsson EC, Tsukayama P, Patel S, Mejía-Bautista M, Sosa-Soto G, Navarrete KM, Calderon M, Cabrera L, Hoyos-Arango W, Bertoli MT,Berg DE, Gilman RH, Dantas G, 2016. Interconnected microbiomes and resistomes in low-income human habitats. Nature, 533(7602):212-6.